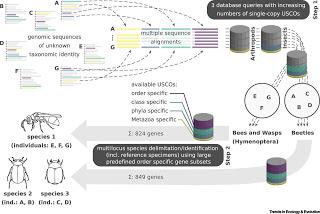
Séquence des Evénements d’Implémentation des Copies Uniques de Gènes de Métazoaires Équivalents sur le Plan Fonctionnel (orthologues) (USCOs) comme Système Universel de Marqueurs pour l’Abornement et l’Identification des Espèces. (…).
La simplicité du séquençage du code-barre ADN a propulsé un système d’identification des espèces universellement applicable à travers les embranchements zoologiques. Cependant, le fait de s’appuyer sur un fragment d’ADN mitochondrial unique présente un certain nombre d’inconvénients qui peuvent induire en erreur l’abornement et l’identification des espèces. L’implémentation de marqueurs nucléaires multiples atténuerait les limites du système actuel de code-barre de l’ADN si ces marqueurs sont applicables chez toutes les espèces, s’ils comprennent suffisamment d’information pour distinguer les espèces les plus proches, et si le séquençage et l’analyse des ces marqueurs peut être automatisée. Alors que les coûts de séquençage continuent de baisser, nous pensons qu’il est maintenant temps d’étendre les usages des codes à barre de l’ADN. Notre argumentation repose sur le fait que presque chaque copie unique universelle de gènes codant pour des protéines nucléaires fournit les caractéristiques souhaitées et pourrait être utilisée pour aborner et identifier de manière fiable les espèces animales. Jonas Eberle, et al, dans Trends in Ecology and Evolution, publication en ligne en avant-première, 15 janvier 2020
Source iconographique, légendaire et rédactionnelle : Science Direct/ Traduction et adaptation : NZ
