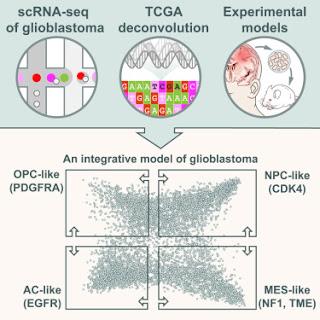
scRNA-seq of glioblastoma = Séquençage d'ARN d'une cellule unique
TCGA deconvolution = Déconvolution à partir de l'Atlas Génomique du Cancer
Experimental models = Modèles expérimentaux
Plusieurs facteurs génétiques, épigénétiques et développementaux président à l’existence du glioblastome, tumeur incurable et peu connue; leur caractérisation précise reste un défi. Ici, nous utilisons une approche intégrative transversale de séquençage d’ARN au niveau d’une cellule unique de 28 tumeurs, d’analyse génétique et de mesure d’expression génique de grande ampleur de 401 spécimens de l’Atlas du Génome du Cancer (TCGA), d’approches fonctionnelles, et du traçage de lignée cellulaire ayant pour origine une cellule unique ; pour fabriquer un modèle unifié d’états cellulaires et de diversité génétique du glioblastome. Nous déterminons ainsi que les cellules malignes de glioblastomes se présentent sous quatre formes principales d’états cellulaires représentant les différents types de cellules nerveuses, sont influencées par le microenvironnement tumoral, et sont douées d'une plasticité. La relative fréquence des cellules représentant chaque stade varie selon les échantillons de glioblastome et est influencée par le nombre de copies de gènes résultant de l’amplification des loci CDK4, EGFR, PDGFRA et par les mutations du locus NF1 ; favorisant chacun un état défini. Notre travail fournit une ébauche descriptive du glioblastome, intégrant les programmes des cellules malignes, leur plasticité, et leur modulation par les divers facteurs génétiques. Cyril Neftel, et al, dans Cell, publication en ligne en avant-première, 18 juillet 2019
Source iconographique, légendaire et rédactionnelle : Science Direct / Traduction et adaptation : NZ
Déconvolution : opération mathématique par laquelle on récupère un objet intégré dans image dégradée par le bruit et le flou.
scRNA : single cell RNA = ARN d’une cellule unique
