La capacité de pister la cinétique de dégradation des protéines à travers le protéome mammifère révèle qu’un échantillon de protéines est de moins en moins susceptible d’être dégradé au fur et à mesure que le temps passe. Les effets d’une variation en nombre de copies de protéines en fonction de la génétique peuvent être prédits par les propriétés de ces protéines.
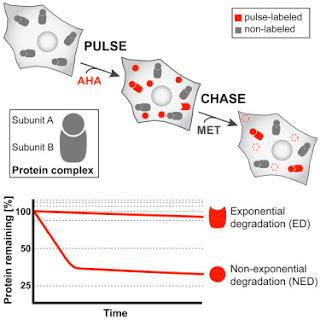
Les protéines récemment synthétisées présentent-elles la même probabilité d’être dégradées que les protéines anciennement synthétisées ? Nous nous sommes posés cette question en utilisant un marquage pulse-chase et la spectrométrie de masse quantitative pour obtenir des profils de dégradation pour des milliers de protéines. Nous trouvons que plus de 10% des protéines subissent une dégradation non-exponentielle. Plus spécifiquement, les protéines sont moins stables au cours des premières heures de leur vie, et se stabilisent avec l’âge. Les profils de dégradation sont conservés, et similaires dans deux types de cellules. Beaucoup de protéines non-exponentiellement dégradées (NED) sont des sous-unités de complexes qui sont produits en quantités super – stoechiométriques par rapport à leurs homologues exponentiellement dégradées (ED). Au sein des complexes, les protéines NED présentent des interfaces d’interaction et s’assemblent de manière plus précoce que les sous-unités ED. L’amplification des gènes codant pour les protéines NED accentue leur dégradation initiale. Les profils de dégradation peuvent toujours prédire l’atténuation des niveaux de protéines dans les cellules aneuploïdes. Prises dans leur ensemble, nos données montrent que la dégradation non exponentielle est commune, conservée, et a des conséquences importantes sur la formation des complexes et la régulation de l’abondance en protéines. Erik McShane, et al, dans Cell, publication en ligne en avant-première, 6 octobre 2016Source iconographique, légendaire et rédactionnelle: Science Direct / Traduction et adaptation : NZ


