 C’est un nouvel outil qui vient d’être développé par ces scientifiques de l’Université de Toronto qui va permettre une meilleure compréhension du rôle jusque-là minimisé des ARNs non codants. Car c’est tout récemment que l’on a appris que notre ADN, dit » poubelle » ou » junk DNA » – car il ne code pas pour des protéines- aurait bien, en réalité, toute sa fonction biologique. Ici, il s’agit bien des ARNs- issus de la transcription de l’ADN- non traduits en protéines. La compréhension de ces molécules reste difficile en raison du manque de technologies permettant d’identifier l’ensemble de leurs éventuelles fonctions. Cette nouvelle méthode présentée dans la revue Molecular Cell, qui décrypte les interactions de ces molécules, va enfin permettre aux scientifiques d’explorer en profondeur la fonction de ces ARNs non codants (ou ARNncs) dans les cellules humaines.
C’est un nouvel outil qui vient d’être développé par ces scientifiques de l’Université de Toronto qui va permettre une meilleure compréhension du rôle jusque-là minimisé des ARNs non codants. Car c’est tout récemment que l’on a appris que notre ADN, dit » poubelle » ou » junk DNA » – car il ne code pas pour des protéines- aurait bien, en réalité, toute sa fonction biologique. Ici, il s’agit bien des ARNs- issus de la transcription de l’ADN- non traduits en protéines. La compréhension de ces molécules reste difficile en raison du manque de technologies permettant d’identifier l’ensemble de leurs éventuelles fonctions. Cette nouvelle méthode présentée dans la revue Molecular Cell, qui décrypte les interactions de ces molécules, va enfin permettre aux scientifiques d’explorer en profondeur la fonction de ces ARNs non codants (ou ARNncs) dans les cellules humaines.
Des 3 milliards de lettres dans le génome humain, seuls 2% constituent les gènes codants pour des protéines. Les gènes sont copiés ou transcrits en molécules d’ARN messager (mRNA) qui fournissent des modèles pour l’assemblage de protéines qui font vivre la cellule. Les 98% restants du génome, dit "ADN poubelle" ont longtemps été privés d’importance fonctionnelle. Le projet Encode mené par 32 instituts de recherche dans le monde a, à travers des milliers d’analyses génétiques de différents tissus, généré une masse énorme de données sur notre génome et commencé à analyser comment le génome impacte les différents types de cellules, les interactions des processus moléculaires sur la production par l’ADN de protéines et des ARN nécessaires à chaque cellule pour fonctionner au sein de notre corps. Encode a ainsi suggéré qu’une très grande partie de l’ADN poubelle, bien que non codant pour des protéines, est bien associé à une fonction biologique. Car cet ADN est capable d’activer et d' » éteindre » des gènes, régulièrement et même à des distances éloignées.
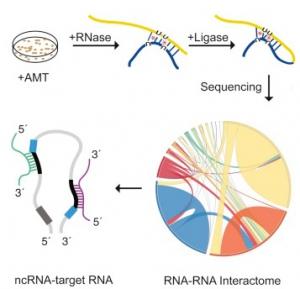
L’équipe de l’Université de Toronto revient sur la fonction de ces ARNs non codants et en particulier, sur leur rôle dans la régulation des gènes. L’équipe rappelle qu’il a déjà été suggéré que certains de ces ARNncs transportent tels des véhicules les mARN autour de la cellule, ou encore fournissent un support à d’autres protéines et aux ARNs leur permettant de se fixer et de faire leur travail. Ce nouvel outil, appelé » LIGR-Seq « , capture les interactions entre les différentes molécules d’ARN. Lorsque deux molécules d’ARN ont des séquences correspondantes, elles s’assemblent et une fois appariées, ces structures d’ARN sont extraites des cellules et analysées par des méthodes de séquençage de pointe, permettant d’identifier précisément les ARNs collés ensemble.
La technologie » LIGR-Seq « va ainsi permettre une nouvelle compréhension de la fonction ARNnc. Pour la première fois les scientifiques devraient pouvoir observer les interactions entre ARNs dans les cellules vivantes et dans toute leur complexité et quelques soient les types, nommés rRNA, tRNA, snRNA, snoRNA, piRNA, miRNA, et lncRNA ARNr, ARNt, snRNA, snoRNA, piRNA, miARN et lncRNA… (et dont les préfixes reflètent la place de l’ARN dans la cellule ou un aspect de leur fonction). La nouvelle technologie pourrait identifier toutes les interactions impliquant toutes les classes d’ARN.
De premières découvertes : L’équipe a découvert de nouveaux rôles pour les petits ARN nucléolaires (snoRNAs) qui guident normalement des modifications chimiques d’autres ARNncs. Ainsi, certains snoRNAs peuvent réguler la stabilité d’un ensemble de » mRNAs » codant pour des protéines. Ainsi, les snoRNAs ont donc une influence directe sur les protéines qui sont fabriquées et sur leurs niveaux de production.
» Une des pointes de l’iceberg « , commentent les chercheurs.
Source: Molecular Cell 19 May 2016 DOI: 10.1016/j.molcel.2016.04.030 Global Mapping of Human RNA-RNA Interactions


