 Parce que multirésistante aux antibiotiques, cette salmonelle, appelée « Salmonella Kentucky », inquiète les scientifiques et en particulier ces chercheurs de l'Institut Pasteur, de l'INRA et de l'Institut de veille sanitaire qui publient les résultats d'une vaste étude internationale dans l'édition du 3 août du Journal of Infectious Diseases. En France aussi, cette bactérie s'est propagée ces 50 dernières années, avec l'apparition de ses différentes résistances. En Europe, aujourd'hui, dans plus de 10% des cas, les patients n'ont pas déclaré de séjour à l'étranger. Les auteurs soulignent donc l'importance d'une surveillance rapprochée de ces bactéries responsables d'infections alimentaires, et la nécessité de rationaliser l'utilisation des antibiotiques dans les filières d'élevage à l'échelle mondiale.
Parce que multirésistante aux antibiotiques, cette salmonelle, appelée « Salmonella Kentucky », inquiète les scientifiques et en particulier ces chercheurs de l'Institut Pasteur, de l'INRA et de l'Institut de veille sanitaire qui publient les résultats d'une vaste étude internationale dans l'édition du 3 août du Journal of Infectious Diseases. En France aussi, cette bactérie s'est propagée ces 50 dernières années, avec l'apparition de ses différentes résistances. En Europe, aujourd'hui, dans plus de 10% des cas, les patients n'ont pas déclaré de séjour à l'étranger. Les auteurs soulignent donc l'importance d'une surveillance rapprochée de ces bactéries responsables d'infections alimentaires, et la nécessité de rationaliser l'utilisation des antibiotiques dans les filières d'élevage à l'échelle mondiale.
Les bactéries du genre Salmonella représentent une des premières causes d'infections alimentaires chez l'homme. Le Centre national de référence (CNR) des Salmonella, à l'Institut Pasteur avait détecté dès 2002, d'un type de salmonelle, chez un petit nombre de voyageurs de retour d'Egypte, du Kenya et de Tanzanie. Cette bactérie, appelée « Salmonella Kentucky », présentait déjà des résistances à de nombreux antibiotiques, notamment aux fluoroquinolones, actuellement l'un des traitements clés des infections sévères à Salmonella.
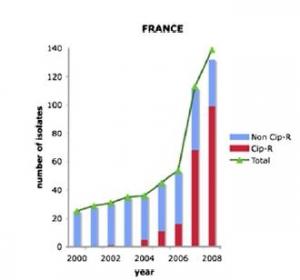
La collecte de données épidémiologiques a permis aux chercheurs de suivre en temps réel la spectaculaire explosion de cette bactérie à partir de 2006 : alors qu'entre 2002 et 2008 on recensait globalement 500 cas pour la France, le Royaume-Uni et le Danemark, 270 cas ont été confirmés pour la France seule entre 2009 et 2010. La zone de contamination, initialement limitée à l'Afrique du Nord-Est et de l'Est, s'est en outre progressivement élargie à l'Afrique du Nord et de l'Ouest, ainsi qu'au Moyen-Orient. Les chercheurs du CNR des Salmonella ont ensuite décrypté les mécanismes de résistance aux antibiotiques et en ont retracé la chronologie. Au début des années 1990, un fragment d'ADN comprenant des gènes de résistance à six molécules, dont certaines étaient déjà largement utilisées à l'époque, s'est intégré dans le chromosome de Salmonella Kentucky. Au milieu des années 1990 est ensuite apparue par mutation la résistance aux quinolones, puis au début des années 2000 celle aux fluoroquinolones.
L'Egypte pourrait être le berceau géographique des trois étapes d'apparition des résistances aux antibiotiques: c'est dans ce pays qu'ont toutes été identifiées pour la première fois les modifications génétiques qui en sont à l'origine. Salmonella Kentucky aurait pu y acquérir le fragment d'ADN responsable des premières résistances par l'intermédiaire des filières aquacoles : le recours massif aux antibiotiques dans ces élevages développés en Egypte dès le début des années 1990 aurait en effet favorisé la sélection des souches bactériennes résistantes à ces antibiotiques. L'explosion récente des cas serait quant à elle liée à la propagation de la bactérie en Afrique dans la filière volaille, grande consommatrice de fluoroquinolones. C'est le cumul de toutes les résistances sur la même souche de Salmonella Kentucky qui serait ainsi à l'origine de l'épidémie actuelle.
La bactérie commence à s'implanter en Europe ce qui inquiète les scientifiques: aujourd'hui, dans plus de 10% des cas, les patients n'ont pas déclaré de séjour à l'étranger. La bactérie commence donc selon toute vraisemblance à s'implanter en Europe, multipliant ainsi le risque d'une contamination de la volaille d'élevage et donc la menace d'une propagation à grande échelle. De plus, les chercheurs de l'Institut Pasteur ont très récemment montré l'existence en Afrique du Nord de quelques souches devenues résistantes aux céphalosporines de troisième génération et aux carbapénèmes.
L'Institut Pasteur rappelle l'importance d'une veille microbiologique en particulier pour les pays du sud, le risque pour la santé humaine de l'utilisation non réglementée des antibiotiques dans les élevages, qui favorise l'apparition et la propagation des gènes de résistance chez des bactéries responsables d'infections alimentaires.
Sources: Communiqué Institut Pasteur, Journal of Infectious Diseases, 3 août 2011, “International spread of an epidemic population of Salmonella enterica serotype Kentucky ST198 resistant to ciprofloxacin”


